加布 鲁迪 2016年12月8日
ExAC CNV被公开发布 ,最近出版publication(作者的意思是2016年,译者注),提供了一整套关于约60K人类exomes的罕见CNV。
这是一篇去年的热门博客。通过这篇博客,可以多了解软件算法设计者的思路。
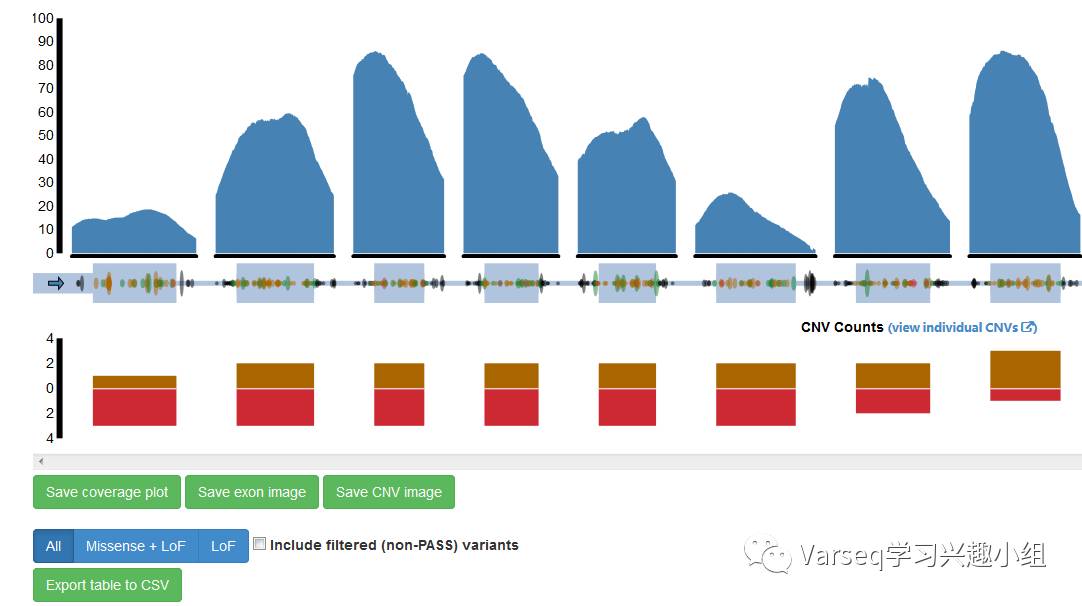
虽然有很多公共CNV数据库,但是这是第一个来自exome的数据,包括非常罕见和非常小的CNV事件。
随着Golden Helix NGS target panel上的CNV识别算法CNV calling algorithm的最新发布,基于NGS的CNV检测对公共数据库的信息利用就更为重要。所以我们继续制作了公共的CNV知识库,现在它们可以作为VarSeq,SVS和GenomeBrowse的注释源。
本文源自
http://blog.goldenhelix.com/grudy/exac-cnvs-the-first-large-scale-public-exome-cnv-variant-set/
针对Exome Coverage Data使用XHMM算法来识别CNV
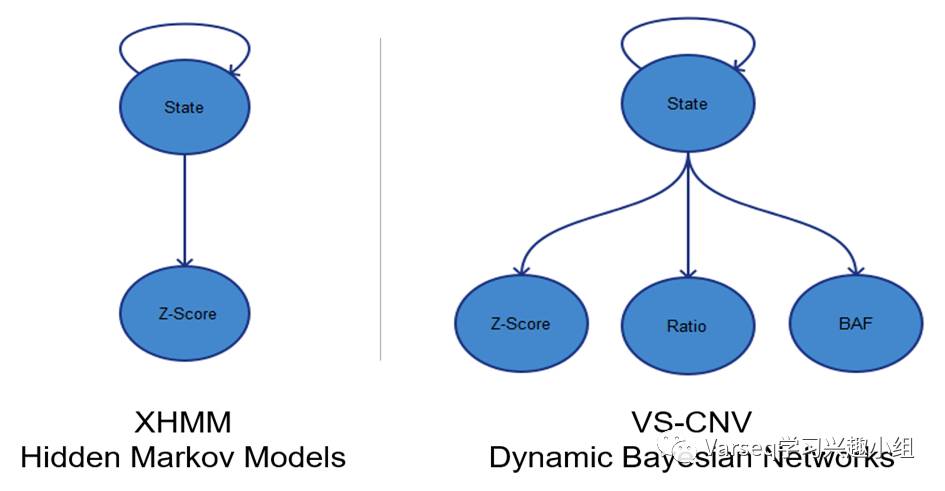
ExAC团队使用XHMM方法在其exome数据中识别CNV。 我们研究了这种方法,可以看到在这样的大型队列研究中很有意义。 XHMM使用PCA进行coverage归一化,这可能需要进行一些微调,以确保把多指标正确的转化为少数几个综合指标(即主成分),获取此参数对于减少足够的噪声但不会丢失信号至关重要。 它还需要相当多的样本才能得到更合理的结果。
相比之下,我们的CNV检出方法-利用参考样本归一化的策略,即使在10个(但推荐的30个)参考样本的情况下也可以工作。
XHMM使用隐马尔可夫模型,根据每个目标区域计算Z-score来检出CNV。 这是我们在文献中看到的最有希望的技术之一,绝对有高度的信心来识别出大型和干净的CNV事件。所以,在我们的方法中,我们扩展了这个概率模型,不仅看Z-score,还考察了目标区域Ratio值和等位变异的频率(见下图)。
由XHMM提供的CNV带有表示XHMM识别置信度的PHREDscale likelihood scores打分。 只有CNV得分> 60分才会被保留。 在解释时,您可能仍然觉得分数小于80也是值得怀疑的。
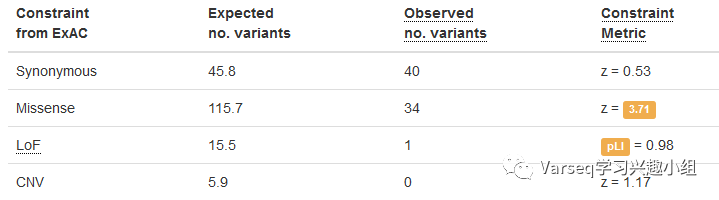
查看XHMM所做的CNV识别得分,会偶尔发现某些分数比较小,但是大部分得分都比较高。对于一些基因,比如PTEN,没有检出到CNV(有很大可能是因为它没有高得分)
ExAC CNVs的结果条都含有哪些内容
GoldenHelix将ExAC HXMM CNV的注释结果也整合到了GenomeBrowse的查询中。如果在软件中导入此结果, 会每个结果条代表了一个CNV的检出。结果条的颜色,deletions为红色(可能被称为杂合或纯合deletions),duplications为蓝色。
Type | # of Calls |
Deletions | 49409 |
Duplications | 77363 |
参照群体,以及个体中找到的CNV,伴随着其quality score分值都会得以呈现。通过GoldenHelix的可视化工具,比如Varseq或者GenomeBrowse进行查看。可以查看具体的CNV状态(deletion/duplication),位置,大小,人群等信息。还可以通过特殊字段,对结果进行筛选来得需要的信息。
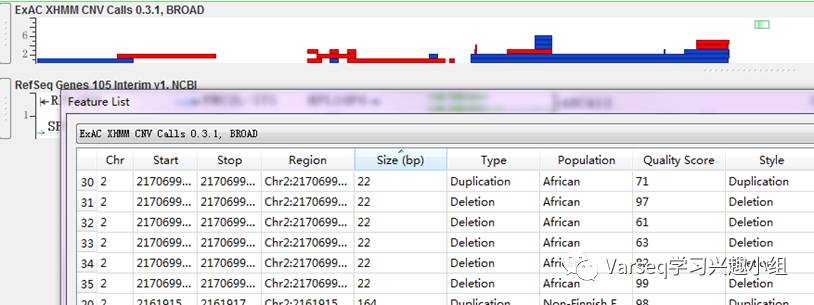
下图是带有个体CNV结果的ExAC CNV结果展示。
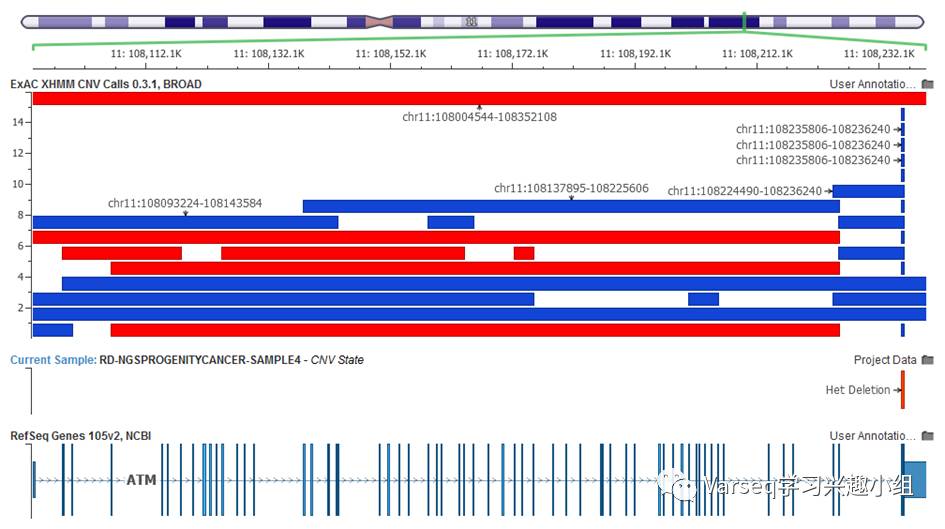
我们样本中的双靶杂合缺失(het deletion)与ExAC 60K exomes中的常见重复(蓝色)发生重叠。
总体来说,结果条可以让您快速浏览CNV检出频率(以及什么类型),并为VarSeq所找到的CNV的可视化提供了绝佳的补充!
关于Gabe Rudy

Gabe Rudy,GHI的产品与工程副总裁,自2002年以来,Gabe在生物信息学和遗传分析的动态和快速变化的领域蓬勃发展。 Gabe在计算机科学家和统计学家的领导下建立强大的产品和提供世界一流的支持,致力于帮助Golden Helix的客户加快研究。当不读书或博客时,Gabe享受户外蒙大拿的生活方式。但最重要的是,Gabe很喜欢和他的儿子,女儿和妻子一起度过时光。参照Gabe在Twitter @gabeinformatics 。




共有条评论 网友评论